Ein Kurs der Regionalen Begabtenförderung Oberfranken – von Clara Hensel
Das erste Treffen unseres Kurses stand unter der Überschrift Chromatographie. Für alle, die nicht wissen was das ist: Bei der Chromatographie nutzt man die unterschiedliche Löslichkeit der Bestandteile eines Farbstoffgemischs, um sie mit Hilfe eines Laufmittels auf einem Träger räumlich zu trennen. Zunächst haben wir Filzstiftpunkte auf Filterpapier untersucht, um die Unterschiede in den Bestandteilen von Stiften unterschiedlicher Hersteller zu untersuchen. Danach haben wir Farbstofflösungen aus Pflanzen gewonnen. Diese wurden auf Kieselgel-DC-Platten aufgetragen (DC = Dünnschichtchromatographie). Bei der Auswertung waren besonders deutlich Carotin bei Karotten und Chlorophyll bei Pflanzen aus dem „DG-Garten“ zu erkennen.

Beim zweiten Treffen haben wir uns mit dem Thema Fluoreszenz und Phosphoreszenz beschäftigt. Unter anderem konnten wir die Chemolumineszenz von Luminoll als blaues Leuchten beobachten, wenn es mit Bluspuren bzw. Hämoglobin in Kontakt kommt. Das eigentlich grüne Chlorophyll hingegen leuchtet unter UV-Beleuchtung intensiv rot.

Mikroskopie war das Thema unseres dritten Termins. Wir begannen damit, einige einfachere Präparate wie Wasserpest und Mundschleimhautzellen anzusehen. Unter anderem beobachteten wir auch an einer roten Zwiebel, wie sich die durch Anthocyane pink gefärbten Vakuolen der Zellen zusammenziehen, wenn man die Zwiebelhaut in eine Salzlösung gibt.
Bei unserem vierten Treffen haben wir eine Exkursion nach Erlangen an die Universität gemacht, wo wir die Möglichkeit hatten, mit Fluoreszenzmikroskopen zu arbeiten. Nach einem kurzen Vortrag zu Themen wie Biolumineszenz, Gentechnik und Drosophila (Fruchtfliegen) ging es dann auch schon ins Labor. Wir mikroskopierten gentechnisch veränderte Drosophila-Larven, die in ihrem Herzmuskel das fluoreszierende GFP-Protein besitzen, das normalerweise nur in biolumineszenten Quallen vorkommt.
 |
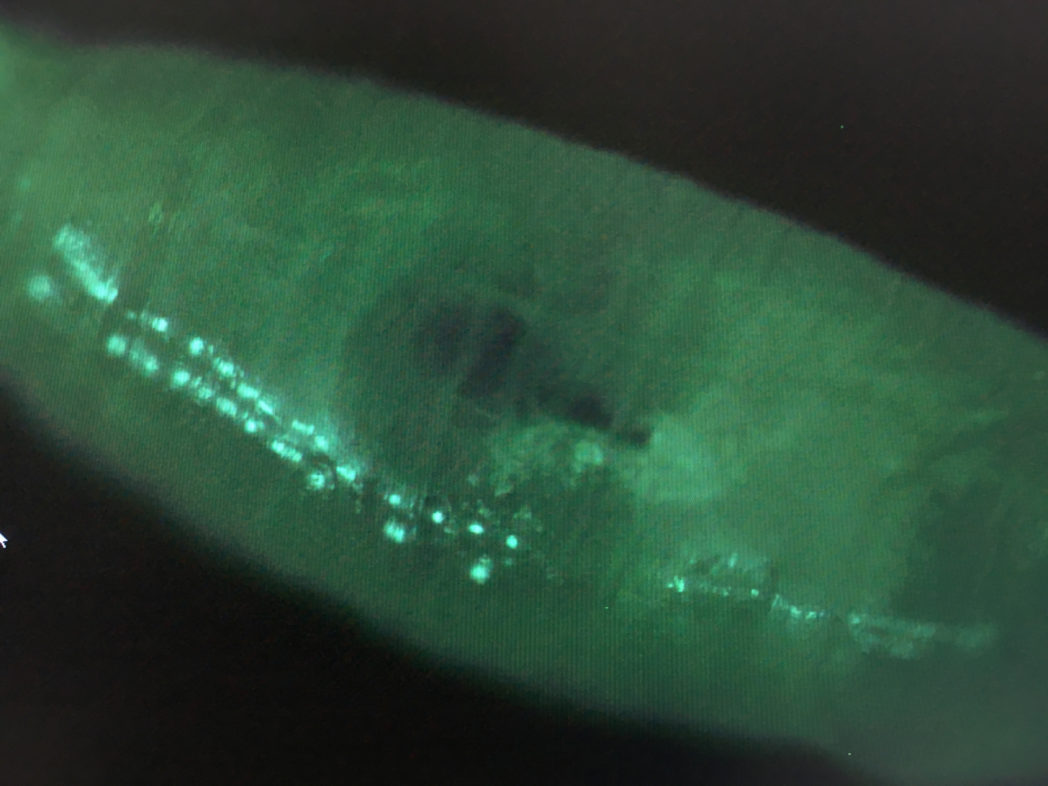 |
So konnten wir im Mikroskop das schlagende Röhrenherz der Insektenlarven sehen, da durch die Anregungsstrahlung nur das grün leuchtende GFP zu sehen war.weshalb ihr Herzmuskel leuchtet. Mit dem Fluriszenzmikroskop kann man ein Präperat noch näher heranholen und noch genauer untersuchen.





































































































































 Nachdem wir im Labor ankamen, bekamen wir zuerst eine Einführung in das wissenschaftliche Arbeiten und vor allem in den Umgang mit Mikropipetten, Zentrifugen, Vortex-Mischern, etc. Dann ging es ans “richtige” Experimentieren…
Nachdem wir im Labor ankamen, bekamen wir zuerst eine Einführung in das wissenschaftliche Arbeiten und vor allem in den Umgang mit Mikropipetten, Zentrifugen, Vortex-Mischern, etc. Dann ging es ans “richtige” Experimentieren…

 Nun, ich denke unter Fingerabdruck kann sich jeder erst einmal etwas vorstellen. Das Muster, das bei einem jeden Menschen einzigartig ist auf den Fingern eben. Viel einzigartiger ist jedoch das Muster der DNA eines jeden Menschen. Durch dieses kann man einen Menschen mit ziemlich hoher Sicherheit identifizieren, weshalb dieses Verfahren beispielsweise in der Kriminalistik zur Überführung von Straftätern verwendet wird, aber auch bei Vaterschaftstest, da jeder Mensch von seinen Eltern einen Teil von deren DNA erbt.
Nun, ich denke unter Fingerabdruck kann sich jeder erst einmal etwas vorstellen. Das Muster, das bei einem jeden Menschen einzigartig ist auf den Fingern eben. Viel einzigartiger ist jedoch das Muster der DNA eines jeden Menschen. Durch dieses kann man einen Menschen mit ziemlich hoher Sicherheit identifizieren, weshalb dieses Verfahren beispielsweise in der Kriminalistik zur Überführung von Straftätern verwendet wird, aber auch bei Vaterschaftstest, da jeder Mensch von seinen Eltern einen Teil von deren DNA erbt. Exkursion zur Landessternwarte Heidelberg
Exkursion zur Landessternwarte Heidelberg de Gebäude, dessen Form an eine Spiralgalaxie erinnert, beherbergt Seminar-, Praktikums- und Unterrichtsräume, Büros und das zentrale Klaus Tschira Auditorium mit einem digitalen Ganzkuppelprojektionssystem. Nach dreistündigem Workshop konnten wir hier eine faszinierende Planetariumsvorführung genießen. Wenn auch der Workshop für uns kaum neue Erkenntnisse brachte (ein Beleg für unsere gute Arbeit während der Kurstreffen), so war die gesamte Exkursion doch für alle Teilnehmer sehr gewinnbringend.
de Gebäude, dessen Form an eine Spiralgalaxie erinnert, beherbergt Seminar-, Praktikums- und Unterrichtsräume, Büros und das zentrale Klaus Tschira Auditorium mit einem digitalen Ganzkuppelprojektionssystem. Nach dreistündigem Workshop konnten wir hier eine faszinierende Planetariumsvorführung genießen. Wenn auch der Workshop für uns kaum neue Erkenntnisse brachte (ein Beleg für unsere gute Arbeit während der Kurstreffen), so war die gesamte Exkursion doch für alle Teilnehmer sehr gewinnbringend.
